我校生物信息科学与技术学院李霞教授研究团队2019年度在肿瘤组学大数据研究中取得了一系列新的研究成果,课题组全面整合和挖掘了肿瘤生物医学大数据资源,从系统层面研究了不同分子导致肿瘤发生发展的分子机制,挖掘新的肿瘤生物标志物,开发新的评估和检测肿瘤治疗反应的大数据平台,相关论文发表在Neuro Oncology(IF:10.091)、Hepatology(IF:14.971)、Nucleic Acids Research(IF:11.147)等国际学术期刊上。
1.系统研究胶质瘤进化性别差异和突变克隆状态
利用高通量的新一代测序数据和单核苷酸多态性芯片数据资源,成功识别了约600例低阶胶质瘤和胶质瘤母细胞瘤病人的突变克隆状态,并在克隆进化水平上解析了性别之间的差异。研究论文《Sex difference of mutation clonality in diffuse glioma evolution》发表在国际著名期刊《Neuro Oncology》(SCI影响因子10.091)上。该研究成果揭示了女性胶质瘤病人携带更高的整体和亚克隆突变负荷,识别了呈现出克隆性性别差异的著名癌基因如CDH18和ATRX,胶质瘤的各个亚型、性别间呈现出突变负荷以及癌基因的克隆性差异,研究还发现临床可靶向的基因突变如MAPK和RTK信号通路突变在性别间存在克隆状态差异,研究证实了在胶质瘤临床上考虑性别这一重要因素的必要性,对临床用药治疗具有一定的指导作用。该文章于2019年2月正式发表。
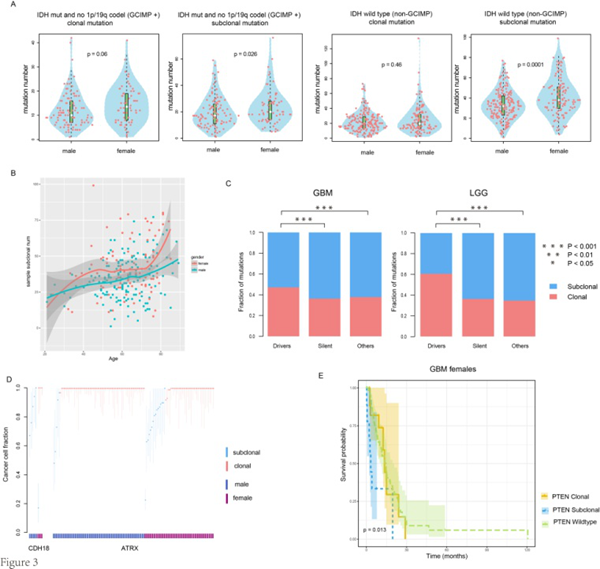
原文链接:https://academic.oup.com/neuro-oncology/article-abstract/21/2/201/5106960
2.系统研究不同状态下RNA结合蛋白与RNA互作
李霞教授团队与德州大学奥斯汀分校伊松副教授团队合作在RNA结合蛋白与RNA互作领域取得了新的突破。研究论文《MERIT: Systematic Analysis and Characterization of Mutational Effect on RNA Interactome Topology》发表在《Hepatology》(SCI影响因子14.971)杂志上。该研究通过探索RBPs的遗传改变模式,发现有害的突变倾向发生在RBPs的表面。通过构建蛋白-RNA互作网络并对网络进行分析发现互作RBP间调控原则,并提出了一种计算方法MERIT来分析不同癌症类型的RBP-基因调控网络。Pan-cancer分析还表明癌细胞选择性的靶向“脆弱性”基因以扰乱参与癌症Hallmark功能的蛋白-RNA互作。肝癌中四对突变扰动的RBP -基因互作被实验证实,它们在细胞增殖中起着至关重要的作用。基于受干扰的RBP和靶基因的表达,还识别了具有不同生存率的三种肝癌亚型。该项工作为研究肝癌中体细胞突变扰动蛋白-RNA的调控网络提供了有价值的资源,对研究癌症中基因型与表型关系提供了有价值的见解。该文章于2019年8月正式发表。
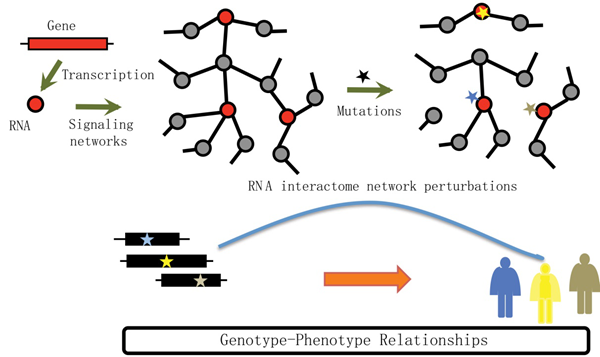
原文链接:https://aasldpubs.onlinelibrary.wiley.com/doi/full/10.1002/hep.30242
3. 人类癌症相关的 lncRNA数据库
本年度在《Nucleic Acids Research》(SCI影响因子11.147)发表了更新版本的数据库lnc2Cancer v2.0,lnc2Cancer数据库目前访问量近12000次,并评为ESI高被引论文。该数据库存储了实验证实的lncRNA与癌症的关系,包括癌症中循环、耐药性及预后相关的lncRNA,以及肿瘤中受到突变、DNA甲基化、miRNA和转录因子调控的lncRNA。最新版的lnc2Cancer数据库中还存储了芯片和新一代测序检测的癌症高通量实验数据,并对癌症根据国际分类标准(ICD-O-3)进行标准化命名。科研人员可以方便的按照癌症原发位置、lncRNA类型、调控机制进行检索和下载数据。该文章于2019年1月正式发表。
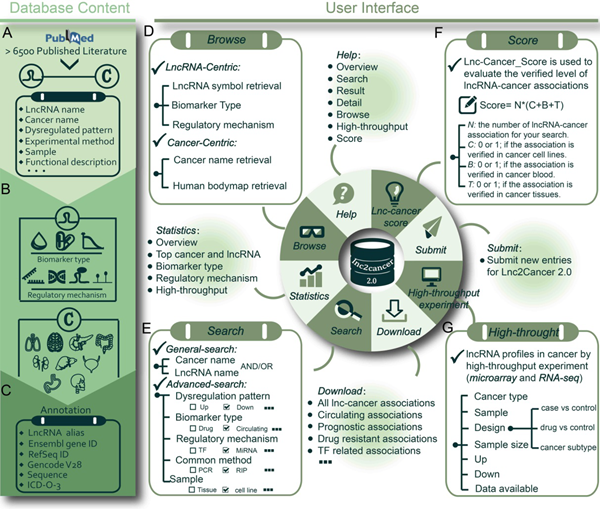
原文链接:https://academic.oup.com/nar/article/47/D1/D1028/5165260
以上研究受到国家自然科学基金等支持。


